La traduction des ARNm
I) Introduction
La traduction permet de convertir l'information génétique contenue dans une séquence nucléotidique d'un ARNm en une séquence d'Acides Aminés qui formera un polypeptide
A la suite de cette traduction, le polypeptide néosynthétisé subira un certain nombre de modifications qui conduiront à la formation d'une protéine fonctionnelle
ARNm----traduction---->polypeptide----maturation---->protéine fonctionnelle
Rappel, pour ne pas confondre
-un polypeptide est la molécule non fonctionnelle
- une protéine est la molécule fonctionnelle
La traduction nécessite énormément d'énergie pour la cellule. Par exemple, E.coli utilise 90% de son énergie pour la traduction

II) Les composants du système de traduction
La traduction d'un ARNm en protéine nécessite 3 composants principaux :\
-l'ARNm : matrice, c'est lui qui porte l'information génétique
- les ribosomes : lecteur de l'information
- les ARNt : font la relation entre les codons et les acides aminés correspondants

1) La matrice (l'ARNm
Un ARNm peut être décomposé en X régions
-une région leader en 5' (appelée "capping" chez les eucaryotes) : elle est non codante, donc non traduite
- un codon initiateur AUG : c'est le premier acide aminé (donc toujours Methionine
- une région codante (suite de codons
- un codon non sens (ou codon STOP) : détermine la fin de la traduction
- une queue en 3' (polyadénylée chez les eucaryotes) : elle est non codante, donc non traduite

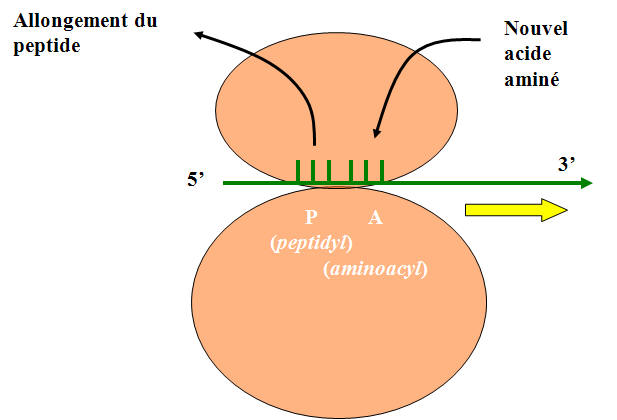
2) Les lecteurs (les ribosomes
Représentent les éléments centraux du système traductionnel
-ils sont indispensables à la reconnaissance entre l'ARNm et les ARNt
- en se positionnant sur la matrice, ils permettent la reconnaissance du codon AUG et donc la mise en place et le maintient du bon cadre de lecture du codon AUG jusqu'à un codon non-sens
- ils vont catalyser la formation de la liaison peptidique (liaison entre les acides aminés
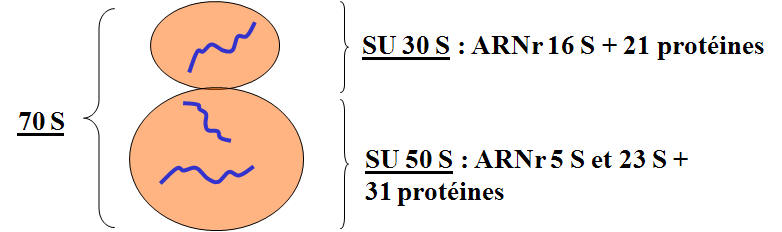
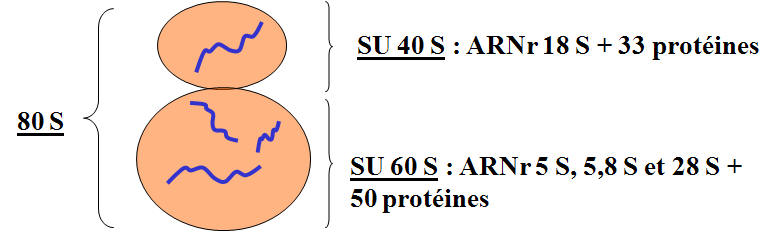
Quelque soit leur origine, ce sont des édifices formés de deux sous-unités capables de s'assembler et de se désassembler. Attention : ce ne sont pas des organites, ils ne sont pas délimités par une membrane, ce sont des particules/assemblages ribonucléoprotéiques
Quand le ribosome se déplace sur l'ARNm, il se déplace dans le sens 5' vers 3'. Lorsqu'il est fixé sur l'ARNm, il recouvre au moins 2 codons (donc 6 acides aminés) qui sont positionnés au niveau de 2 sites particuliers :\
-A (aminoacyle) : position où le nouvel acide aminé prend place
- P (peptidyle) : position où le peptide s'allonge
Structure des ribosomes procaryotes
Structure des ribosomes eucaryotes
3) Les adaptateurs (les ARNt
1. Structure des ARNt
Rôle : assurer le relai entre les codons de l'ARNm et les acides aminés spécifiés par ces codons. Un ARNt doit donc avoir une double fonction de reconnaissance : pour le codon d'une part, et de l'acide aminé correspondant à ce codon d'autre part
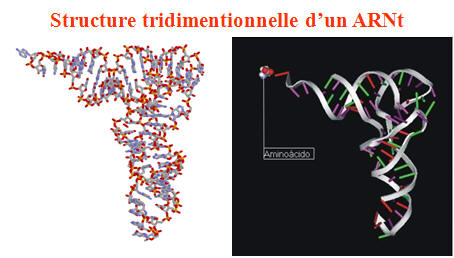
Ils possèdent une organisation structurale très particulière : il en existe une 50aine différents, certains reconnaissant plusieurs codons
Leur structure en feuille de trèfle est dûe à des autoappareillements au sein de la molécule
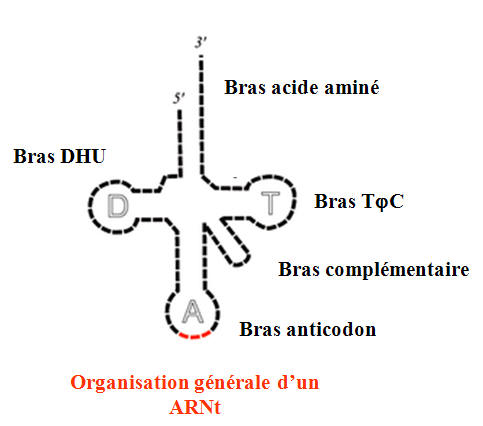
Ils sont constitués de plusieurs parties
-le bras AA : formé par l'auto appareillement des deux extrémités de la molécule. C'est à ce niveau que sera fixé l'Acide Aminé correspondant (sur l'extrémité 3'). Se termine donc toujours par CCA
- le bras DHU : caractérisé par la présence de bases particulières : les di-hydro-uridyles (DHU
- le bras TΦC : présence de ribothymidine et de pseudo-uridyle
- le bras complémentaire : optionnel
- le bras anticodon : va porter une séquence de 3 nucléotides complémentaires des codons de l'ARNm
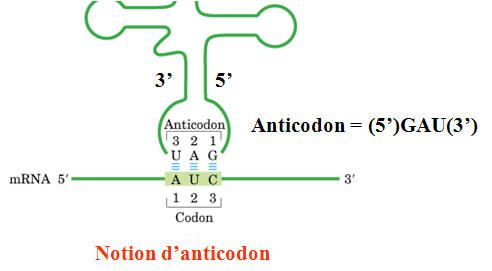
2. Charge des ARNt par les acides aminés
Les ARNt existent sous 2 formes dans une cellule
- forme non chargée = pas d'acide aminé fixé
- forme chargée : un acide aminé fixé
Par convention, les différents types d'ARNt sont nommés en faisant référence à l'acide aminé qu'ils reconnaissent
Exemple
- Non chargés
¤ Leucine =ARN t Leu
¤ Asparagine =ARN tAsp
- Chargés
¤ Leu - ARN t Leu
¤ Asp - ARN tAsp
La charge


III) Les étapes de la traduction
La traduction se divise en 3 étapes
-l'initiation
- l'élongation
- la terminaison
1) L'initiation de la traduction
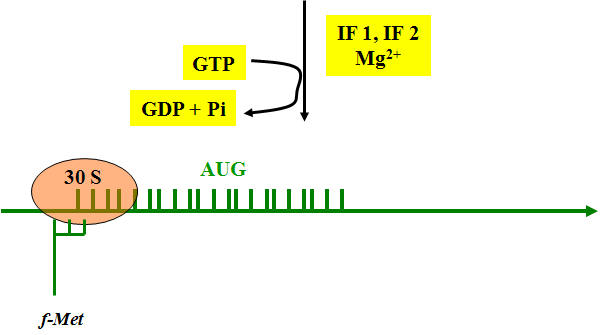
3 étapes nécessitant 3 facteurs d'initiation (IF = Initiation Factor), du GTP et du MG 2
1. Fixation de la petite sous unité ribosomique sur la région leader 5' de l'ARNm

Chez les procaryotes, la petite sous unité se place sur une séquence particulière : la séquence de Shine et Dalgarno (séquence 16S et séquence S et D sont complémentaires)
Chez les eucaryotes, il n'y a pas de séquence comparable. On pense que c'est la séquence de la coiffe qui est responsable de l'initiation
2. Positionnement du 1er ARNt sur la petite sous unité ribosomique
3. Déplacement du complexe sur le codon AUG. Fixation de la grosse sous unité et positionnement de l'ARNt au site P. Relargage des IF
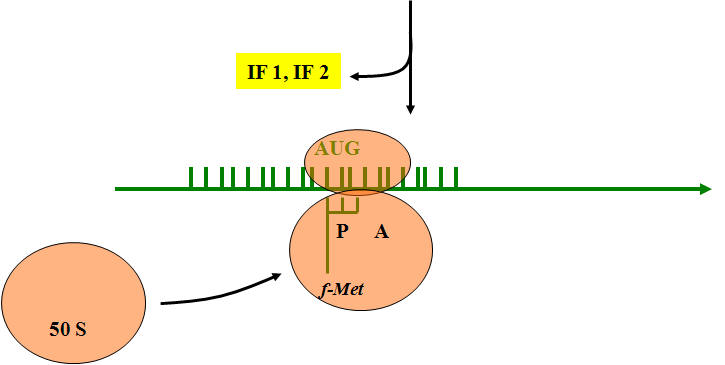
2)
La phase d'élongation
Nécessite 3 facteurs d'élongation (EF = Elongation Factor), du GTP et du MG 2+
1. Fixation de l'ARNt chargé sur le site A du ribosome
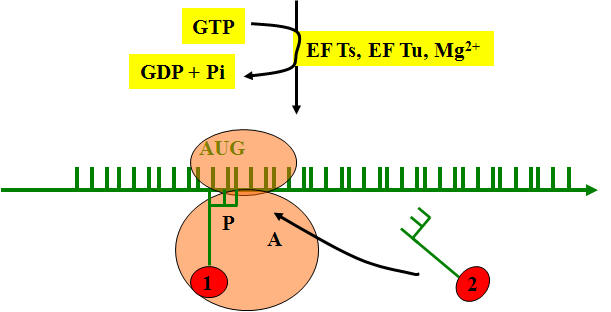
2. Formation de la liaison peptidique et libération du site P
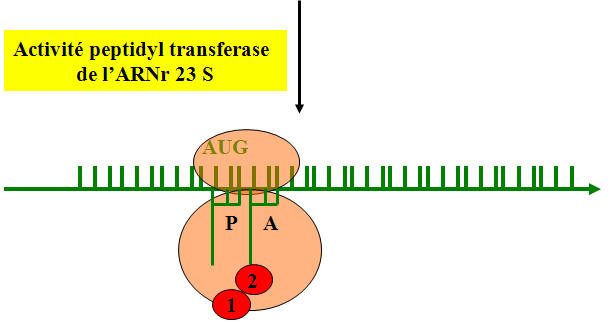
3) La phase de terminaison
Nécessite un codon STOP (UAA; ou UAG; ou UGA), des facteurs de terminaison = Facteur R (Release Factor
1. Blocage de la translocation
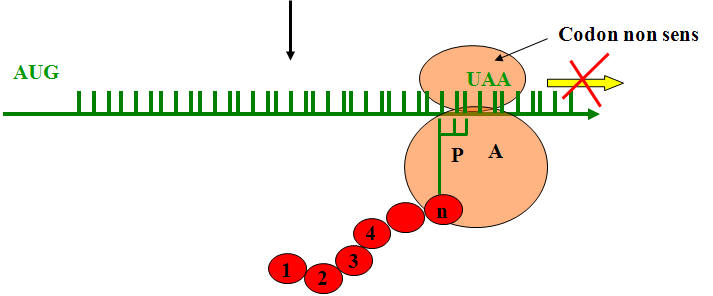
2. Relargage de la chaine polypeptidique

3. Séparation des sous unités ribosomiques
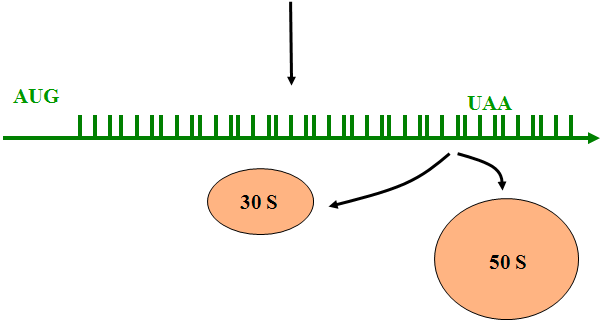
Ainsi se termine la traduction